딥러닝관련 분야에서 시계열 분류를 어떤 방식으로 진행할지 항상 문제점으로 화두가 되어 왔습니다.
예전에는 Recurrent Neural Networks(RNN)이라는 방식의 LSTM, GRU 방식이 많이 사용되었지만 훈련하기 어렵고 정확한 결과가 산출되지 않는 문제점들이 나오기 시작하면서 이미지 처리 분야에서 사용되고 있는 CNN 기반의 알고리즘들이 시계열 데이터 처리 분야에 많이 사용되기 시작하고 있습니다.
https://twobeach.tistory.com/47?category=976038
[python] 시계열 데이터를 Recurrence Plot을 이용하여 Image로 표현하기
이미지 인식 분야에서 사람을 능가하는 성능을 보여주는 딥러닝 모델인 CNN을 시계열 데이터에도 적용하기 위해 다양한 시도와 접근들이 이뤄지고 있었고 가장 직관적으로 생각할 수 있는 접근
twobeach.tistory.com
이번에 소개드릴 알고리즘은 앞서 다루었던 RP 알고리즘과 같은 시계열 데이터 이미지화 알고리즘입니다.
Gramian Angular Field의 경우 그램 매트릭스라는 방식을 사용하고 있는데 이 그램 매트릭스는 선형 대수학과 기하학에 유용한 도구입니다. 무엇보다 벡터의 선형 종속성을 계산하는 곳에 자주 사용되고 있습니다.
그럼 왜 그램 매트릭스 방식을 사용하냐면 그램 매트릭의 경우 시간적 종속성을 유지할 수 있고 아래의 GIF 사진처럼 왼쪽 위에서 오른쪽 아래로 이동함에 따라 시간이 증가하기 때문에 시간 차원의 행력 기하학으로 인코딩할 수 있기때문에 Gramian Angular Field에서 사용되고 있습니다.

Gram Matrix를 사용하기 전에 시계열을 2차원 이상의 공간으로 인코딩해야 하는데 이를 위해 정보를 잃지 않도록 1D 시계열과 2D 공간 간에 bijective mapping 을 구성하게 됩니다.
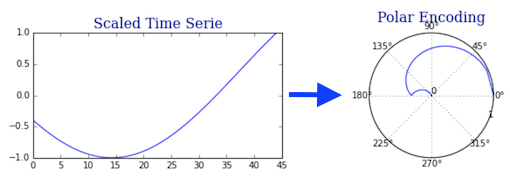
이 인코딩 방식은 polar coordinates transformations 에서 크게 영감을 얻었다고 하며, 이 경우 반지름 좌표는 시간을 표현합니다.
1단계: 최소-최대 스케일러를 사용하여 시리즈를 [-1, 1]로 확장
Min-Max 스케일러와 결합하여 극좌표 인코딩은 bijective이며 arccos function bijective를 사용합니다 (다음 단계 참조).
2단계: 스케일링된 시계열을 "극좌표"로 변환
시계열 값 과 해당 타임스탬프의 두 가지 수량을 고려해야 합니다 . 이 두 변수는 각각 각도 와 반경 으로 표현됩니다 .
시계열이 N 개의 타임스탬프 t 와 해당 값 x 로 구성되어 있다고 가정하면 다음과 같습니다.
- 각도는 arccos(x)를 사용하여 계산됩니다 . [0, ∏] 안에 있습니다.
- 반경 변수는 먼저 [0, 1] 간격을 N개의 동일한 부분 으로 나눕니다 . 따라서 우리는 N+1개의 구분 점 {0, ..., 1}을 얻습니다 . 그런 다음 0을 버리고 이 점을 시계열에 연속적으로 연결합니다.
수학적으로는 다음과 같이 번역됩니다.
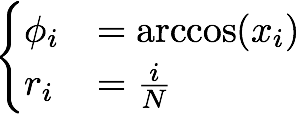
https://medium.com/analytics-vidhya/encoding-time-series-as-images-b043becbdbf3
Encoding time series as images
Gramian Angular Field Imaging
medium.com
이론적인 부분은 위의 내용을 참고하였으며, 자세한 내용은 위의 포스팅를 참조하시면 됩니다.
이런 복잡한 과정을 통해 이미지화가 진행이 되며, 코드적으로 사용하실때에는 간단하게 라이브러리를 통해서 코딩하실수 있습니다.
기본 예제코드
in:
from pyts.datasets import load_gunpoint
from pyts.image import GramianAngularField
X, _, _, _ = load_gunpoint(return_X_y=True)
transformer = GramianAngularField()
X_new = transformer.transform(X)
X_new.shapeout:
(50, 150, 150)- 각도 합을 코사인이나 사인값으로 변형
- 각도 합의 코사인값을 계산 (the cosine of the sum of the angles for the Gramian Angular Summation Field (GASF)
- 사인값을 계산 (the sine of the difference of the angles for the Gramian Angular Difference Field (GADF).)
in:
# Show the results for the first time series
import matplotlib.pyplot as plt
from mpl_toolkits.axes_grid1 import ImageGrid
gasf = GramianAngularField(image_size=24, method='summation')
X_gasf = gasf.fit_transform(X)
gadf = GramianAngularField(image_size=24, method='difference')
X_gadf = gadf.fit_transform(X)
# Show the images for the first time series
fig = plt.figure(figsize=(8, 4))
grid = ImageGrid(fig, 111,
nrows_ncols=(1, 2),
axes_pad=0.15,
share_all=True,
cbar_location="right",
cbar_mode="single",
cbar_size="7%",
cbar_pad=0.3,
)
images = [X_gasf[0], X_gadf[0]]
titles = ['Summation', 'Difference']
for image, title, ax in zip(images, titles, grid):
im = ax.imshow(image, cmap='rainbow', origin='lower')
ax.set_title(title, fontdict={'fontsize': 12})
ax.cax.colorbar(im)
ax.cax.toggle_label(True)
plt.suptitle('Gramian Angular Fields', y=0.98, fontsize=16)
plt.show()out:

in:
from pyts.image import GramianAngularField
from pyts.datasets import load_gunpoint
from sklearn.pipeline import make_pipeline
from sklearn.linear_model import LogisticRegression
X_train, X_test, y_train, y_test = load_gunpoint(return_X_y=True)
gaf = GramianAngularField(flatten=True)
logistic = LogisticRegression(solver='liblinear')
clf = make_pipeline(gaf, logistic)
clf.fit(X_train, y_train)
clf.score(X_test, y_test)out:
0.9733333333333334
pyts의 나와있는 예제를 기준으로 작성되었고, 실제 제가 가진 데이터를 주기마다 이미지로 변환 되게 코딩을 하였습니다.
from pyts.datasets import load_gunpoint
from pyts.image import GramianAngularField
#from pyts.image import GASF, GADF
from scipy.signal import find_peaks
from scipy.misc import electrocardiogram
from scipy.spatial.distance import pdist, squareform
from pyts.image import MarkovTransitionField
import pylab as plt
import pandas as pd
import numpy as np
import math
import matplotlib.pyplot as plt
from mpl_toolkits.axes_grid1 import ImageGrid
from sklearn.preprocessing import MinMaxScalerin:
data_frame_TNT = pd.read_csv("Concentration_IMS/RDX_test_10ng.csv")
print(data_frame_TNT)
graph_data_or = data_frame_TNT['test_1']
#peaks, properties = find_peaks(x, height=(None, 7600000))
#print(f'Index of each peaks : {peaks} ')
#print(f'Index of each peaks : {properties["peak_heights"]} ')
print(math.ceil(len(graph_data_or)/4455)-1)
graph_num=[]
peak_num_count=[]
sum_where_min_peak=[]
a = []
b = []
#for i in range(math.ceil(len(graph_data_or)/4455)-1):
for i in range(0,3526):
graph_num = graph_data_or[(i*4455):((i+1)*4455)]
#print(graph_num)
peaks, properties = find_peaks(graph_num, height=(None, 8400000))
peaks_num = peaks # 피크의 위치들
peaks_value = properties["peak_heights"] # 피크의 위치들의 값
min_peak_value = min(peaks_value) # 피크의 값중 가장 값
min_peak_find = np.where(peaks_value==min_peak_value) # 탐색 된 가장 작은 피크값의 위치 값
print(peaks_num[min_peak_find])
#print(min_peak_value)
where_min_peak = peaks_num[min_peak_find] #가장 작은 피크값들 위치 모음
if i > 0:
#sum_where_min_peak = where_min_peak + sum_where_min_peak # 가장 작은
sum_where_min_peak = where_min_peak
a= np.array(sum_where_min_peak)
else:
sum_where_min_peak = where_min_peak
a= np.array(sum_where_min_peak)
#a= np.array(where_min_peak) #peak값 위치모음 numpy배열에 저장
#print(a)
b= np.array(i*4455) #peak 위치값의 정확한 위치를 파악하기 위해 더해주는 값
#print(b)
peak_num_count = np.append(peak_num_count,a+b) #작은 피크들 위치 값 배열에 저장
print(peak_num_count)
graph_num.plot(alpha = 0.5)
plt.scatter((i*4455)+peaks_num[min_peak_find], min_peak_value, c = 'r') # 피크들 그래프 상에 점의 형식으로 띄우는 것
print(peak_num_count)out:
6326
[355]
[355.]
[354]
[ 355. 4809.]
[359]
[ 355. 4809. 9269.]
[417]
[ 355. 4809. 9269. 13782.]
[357]
[ 355. 4809. 9269. 13782. 18177.]
[355]
[ 355. 4809. 9269. 13782. 18177. 22630.]
[355]
[ 355. 4809. 9269. 13782. 18177. 22630. 27085.]
[351]
[ 355. 4809. 9269. 13782. 18177. 22630. 27085. 31536.]
[357]
[ 355. 4809. 9269. 13782. 18177. 22630. 27085. 31536. 35997.]
[359]in:
data_frame_TNT_Cut_main = pd.read_csv("Concentration_IMS/RDX_test_10ng.csv")
print(data_frame_TNT_Cut_main)
cnt = 1
nsample = 0
Cut_data_main = data_frame_TNT_Cut_main[['test_1']]
peak_num_count_=[]
for c in range(len(peak_num_count)):
peak_num_count_.append(int(peak_num_count[c]))
#print(peak_num_count_)
peak_num_count_.append(0)
#for c in range(math.ceil(len(Cut_data_main)/4455)-1):
for c in range(0,3526):
nc1 = peak_num_count_[c-1]
nc2 = peak_num_count_[c]
if nc1 != nc2:
gg = Cut_data_main[nc1:nc2]
#print(len(gg))
#print(gg)
#cc = int(math.ceil(len(gg)))
#print(cc)
#nsample = cc
gg_=np.array(gg)
save_data=pd.DataFrame(gg_)
save_data.to_csv('peak_con/rdx/10n/RDX_10ng_%d.csv'% (cnt), encoding='utf-8', index=False)
'''
scaler = MinMaxScaler()
scaler_fit = scaler.fit(gg)
scaler_data = scaler.transform(gg)
gasf = GramianAngularField(image_size=1, method='summation')
X_gasf = gasf.fit_transform(gg_new)
print(X_gasf)
gadf = GramianAngularField(image_size=1, method='difference')
X_gadf = gadf.fit_transform(gg_new)
gaf = GramianAngularField(image_size=1)
im_gg = gaf.fit_transform(scaler_data)
print(im_gg)
plt.imshow(im_gg[0])
plt.show()
'''
#plt.imshow(gg_1)
#plt.savefig('Concentration_IMS/RDX_10ng/RDX_10ng_%d.png'% (c))
cnt = cnt + 1out:
test_1
0 8384771
1 8365063
2 8320657
3 8308695
4 8315119
... ...
28184919 8382683
28184920 8407849
28184921 8390387
28184922 8344001
28184923 8345853
[28184924 rows x 1 columns]in:
cnt_=1
for c in range(0,3526):
data_imaging = pd.read_csv("peak_con/rdx/10n/RDX_10ng_%d.csv"% (cnt_))
data_imaging_gg = data_imaging[['0']]
data_imaging_gg = np.array(data_imaging_gg)
#print(data_imaging_gg)
nsample, nx = data_imaging_gg.shape
data_imaging_gg_new = data_imaging_gg.reshape((1,nsample))
#data_imaging_gg_n = GramianAngularField().transform(data_imaging_gg_new)
print(data_imaging_gg_new.shape)
#print(data_imaging_gg_n)
#gaf = GramianAngularField(image_size=nsample)
if nsample >= 100:
#im_gg = gaf.fit_transform(data_imaging_gg_new)
gasf = GramianAngularField(image_size=100, method='summation')
X_gasf = gasf.fit_transform(data_imaging_gg_new)
gadf = GramianAngularField(image_size=100, method='difference')
X_gadf = gadf.fit_transform(data_imaging_gg_new)
fig = plt.figure(figsize=(8,4))
grid = ImageGrid(fig, 111,
nrows_ncols=(1,2),
axes_pad=0.15,
share_all=True,
cbar_location="right",
cbar_mode="single",
cbar_size="7%",
cbar_pad=0.3,
)
images = [X_gasf[0], X_gadf[0]]
titles = ['Summation', 'Difference']
for image, title, ax in zip(images, titles, grid):
im = ax.imshow(image, cmap='rainbow', origin='lower')
ax.set_title(title, fontdict={'fontsize': 12})
ax.cax.colorbar(im)
ax.cax.toggle_label(True)
plt.suptitle('Gramian Angular Fields', y=0.98, fontsize=16)
plt.savefig('peak_con/rdx/10n/img/RDX_10ng_%d.png'% (cnt_))
plt.show()
'''
print(im_gg.shape)
plt.imshow(im_gg[0])
plt.show()
plt.savefig('peak_con/rdx/50n/img/RDX_50ng_%d.png'% (cnt_))
'''
cnt_=cnt_ + 1out:

이 알고리즘을 cnn에 적용했을 때 좋은 결과가 있다는 점을 이용해서 다음에 따로 실험을 진행해볼 예정입니다.
'코딩 > python - 코드' 카테고리의 다른 글
| SciPy와 matplotlib를 이용한 시계열 데이터 피크 검출 (0) | 2021.07.09 |
|---|---|
| [python] 시계열 데이터를 Recurrence Plot을 이용하여 Image로 표현하기 (0) | 2021.04.06 |
| [python] file 소켓을 통한 txt파일 전송 서버->클라이언트 (0) | 2020.11.10 |